感谢关注,一起来学习生信干货。
今日代码实现
玩转RunUMAP参数,修改UMAP图cluster的聚类形状
编者注
老赵最早做单细胞分析的时候,读别人的文章,有时候会很困惑:为什么别人家的UMAP图那么圆润,而自己的UMAP图枝枝丫丫,显得丑陋不堪。后面做多了单细胞,在假设没有恶意删除离群细胞的情况下,自己也摸索出了一些修图调参的技巧,在这里以飨读者。
读取数据,默认参数 PCA 1:30
library(Seurat)
library(Matrix)
library(ggplot2)
library(dplyr)
library(tidyr)
pbmc10k <- readRDS("/Users/zhao/Desktop/demon_pbmc10k/pbmc10k.rds")
pbmc10k <- RunUMAP(pbmc10k, dims = 1:30,reduction = 'pca')
DimPlot(pbmc10k,group.by = 'celltype',label = T)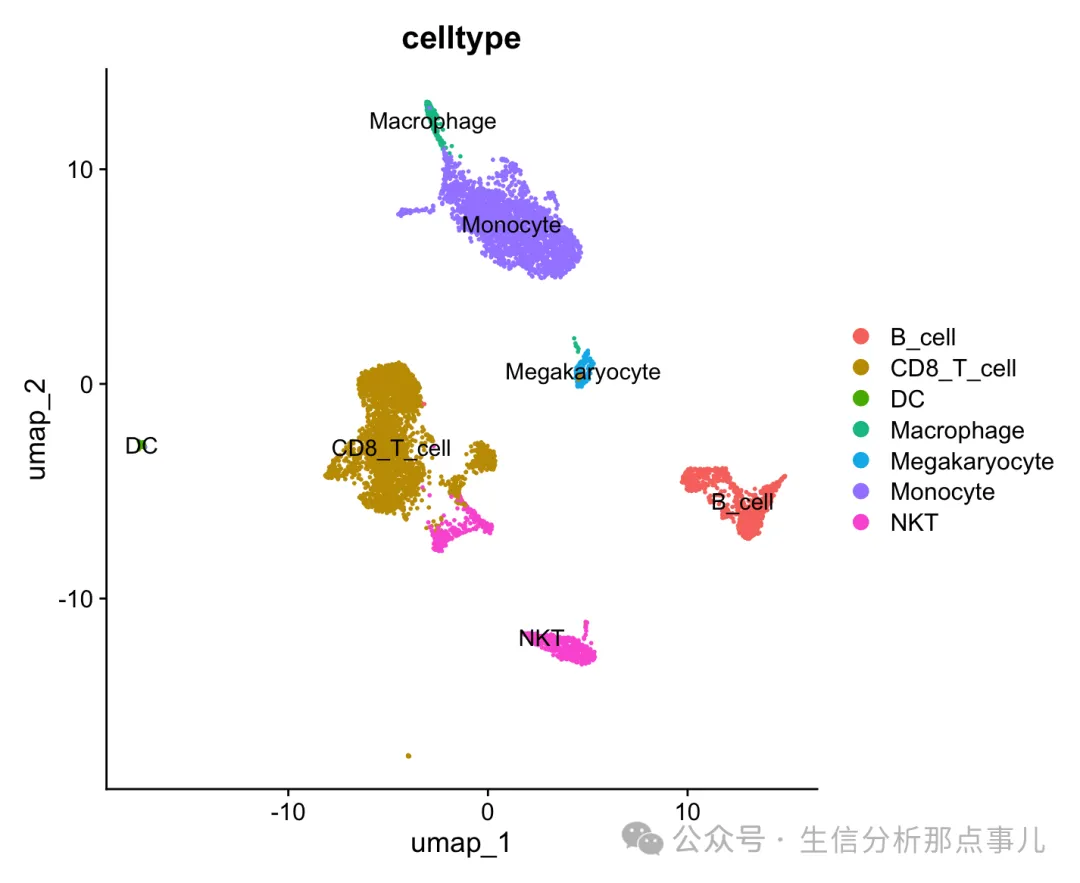
可以看到在利用dim1:30 PCA的时候,NKT有明显两群。
1. 可调参数之PCA dims
# 探究PCA的dims
# 定义不同的 dims 范围
dims_list <- list(1:10, 1:20, 1:40, 1:50)
plot_list <- list()
# 循环运行 RunUMAP 并绘制图片
for (i in seq_along(dims_list)) {
dims <- dims_list[[i]]
# 运行 UMAP
pbmc10k <- RunUMAP(pbmc10k, dims = dims, reduction = 'pca')
# 绘制 UMAP 图
plot <- DimPlot(pbmc10k,group.by = 'celltype', pt.size = 0.5, label = TRUE)
plot <- plot + ggtitle(paste("Dims:", paste(head(dims, 1), ":", tail(dims, 1))))
# 将图片添加到列表中
plot_list[[i]] <- plot
}
# 使用 patchwork 的 wrap_plots 函数将所有图片组合成两列
final_plot <- wrap_plots(plot_list, ncol = 2) & theme(plot.title = element_text(size = 16, face = "bold"))
final_plot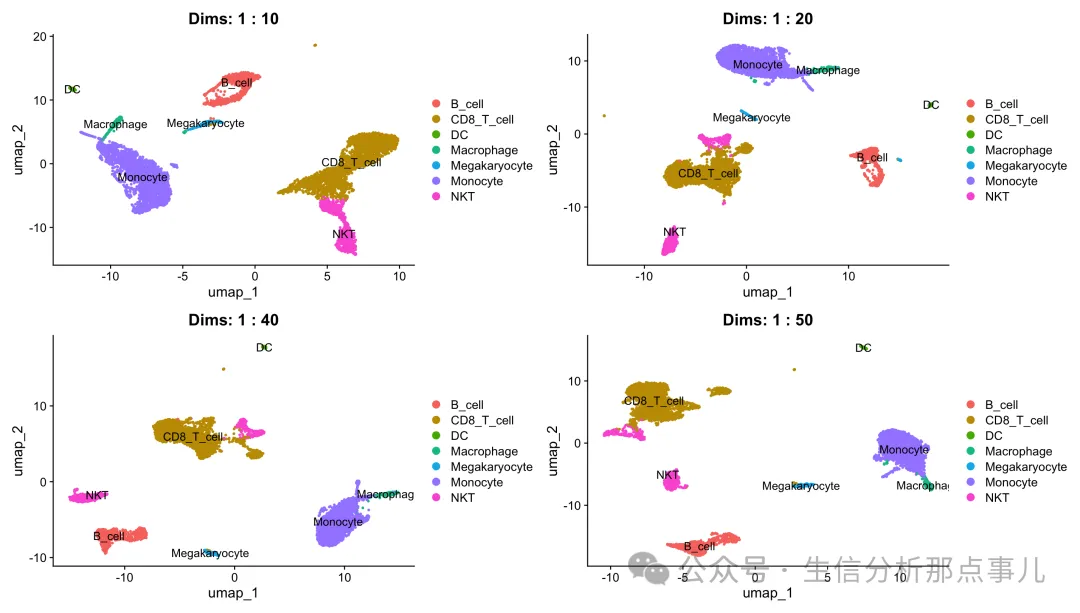 可以看到PCA纬度对聚类的影响:
可以看到PCA纬度对聚类的影响:
在1:10的时候NKT的两群被合并了;而1:20起,NKT会分成两群。
同样在1:10的时候CD8 T是一群,但是在增大纬度后,会逐渐分成两群。
结论就是:增加PCA纬度,会让亚群分的更开。
2. 可调参数之 n.neighbors

# 探究neighbors的数量
neighbor_list <- list(5, 20, 30, 50)
plot_list <- list()
# 循环运行 RunUMAP 并绘制图片
for (i in seq_along(neighbor_list)) {
neighbor <- neighbor_list[[i]]
# 运行 UMAP
pbmc10k <- RunUMAP(pbmc10k, dims = 1:30, n.neighbors = neighbor ,reduction = 'pca')
# 绘制 UMAP 图
plot <- DimPlot(pbmc10k,group.by = 'celltype', pt.size = 0.5, label = TRUE)
plot <- plot + ggtitle(paste("n.neighbors:", neighbor))
# 将图片添加到列表中
plot_list[[i]] <- plot
}
# 使用 patchwork 的 wrap_plots 函数将所有图片组合成两列
final_plot <- wrap_plots(plot_list, ncol = 2) & theme(plot.title = element_text(size = 16, face = "bold"))
final_plot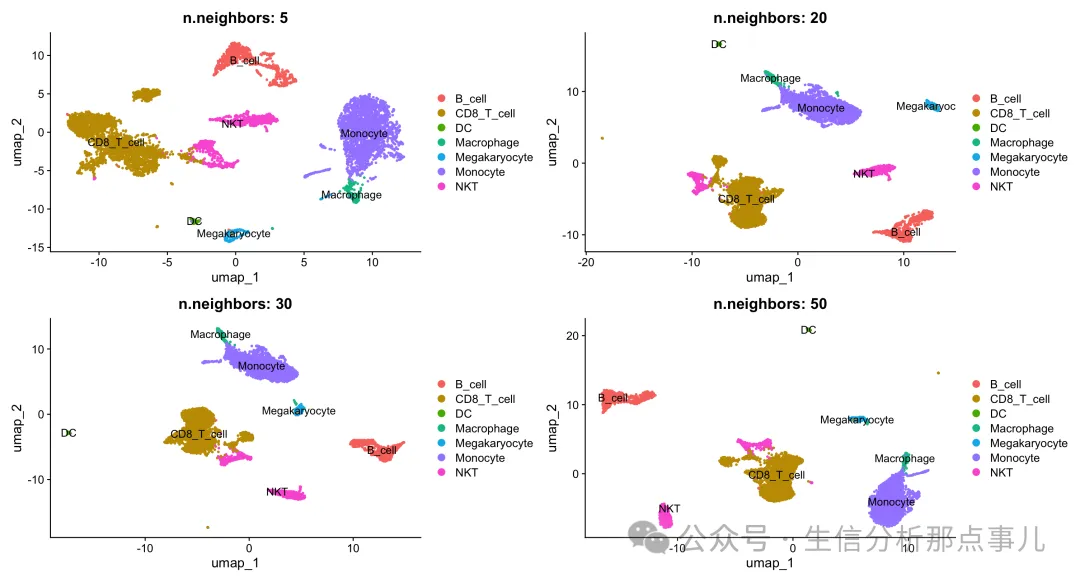 由上图可见:neighbors不影响亚群的区分,但是影响聚类的整体圆润度,以B细胞为例,n.neighbor等于5的时候,枝枝丫丫;但是到50的时候就显得圆润舒服很多。
由上图可见:neighbors不影响亚群的区分,但是影响聚类的整体圆润度,以B细胞为例,n.neighbor等于5的时候,枝枝丫丫;但是到50的时候就显得圆润舒服很多。

3. 可调参数之 min.dist


# 探究min.dis
min.dis_list <- list(0.001,0.01,0.1,0.5)
plot_list <- list()
# 循环运行 RunUMAP 并绘制图片
for (i in seq_along(min.dis_list)) {
min_dist <- min.dis_list[[i]]
# 运行 UMAP
pbmc10k <- RunUMAP(pbmc10k, dims = 1:30 ,reduction = 'pca',min.dist = min_dist)
# 绘制 UMAP 图
plot <- DimPlot(pbmc10k,group.by = 'celltype', pt.size = 0.5, label = TRUE)
plot <- plot + ggtitle(paste("min.dist:", min_dist))
# 将图片添加到列表中
plot_list[[i]] <- plot
}
# 使用 patchwork 的 wrap_plots 函数将所有图片组合成两列
final_plot <- wrap_plots(plot_list, ncol = 2) & theme(plot.title = element_text(size = 16, face = "bold"))
final_plot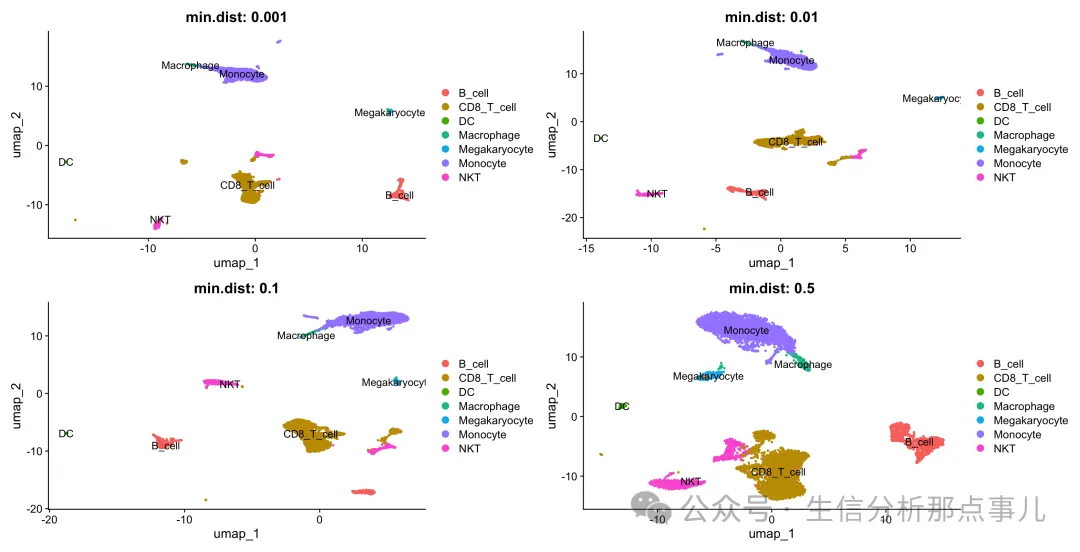 从上图可以看到,min.dist越小,cluster越稀碎;越大,cluster越聚集。如果离散点很多,就增加min.dist到0.5, 同时cluster会显得更胖。
从上图可以看到,min.dist越小,cluster越稀碎;越大,cluster越聚集。如果离散点很多,就增加min.dist到0.5, 同时cluster会显得更胖。
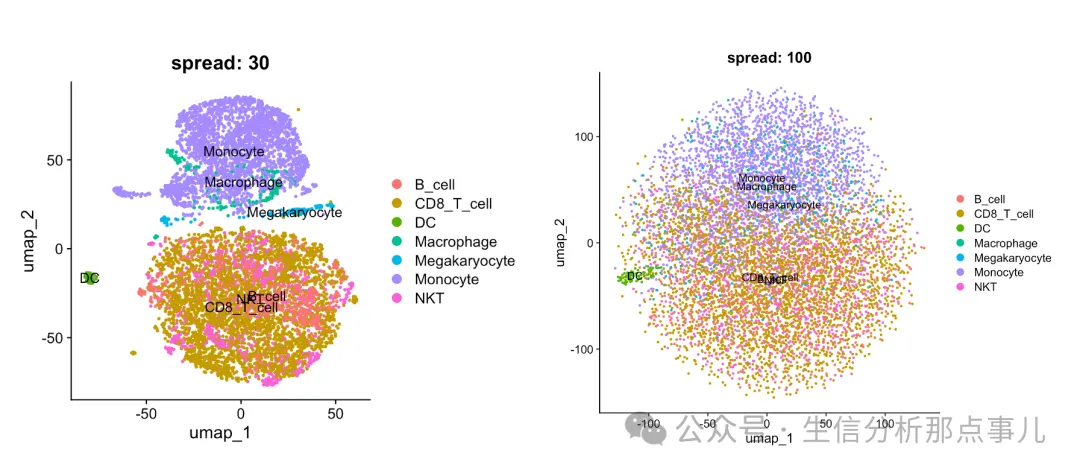
4. 可调参数之 spread

# 探究spread
spread_list <- list(1,5,10,20)
plot_list <- list()
# 循环运行 RunUMAP 并绘制图片
for (i in seq_along(spread_list)) {
spread <- spread_list[[i]]
# 运行 UMAP
pbmc10k <- RunUMAP(pbmc10k, dims = 1:30 ,reduction = 'pca',spread = spread)
# 绘制 UMAP 图
plot <- DimPlot(pbmc10k,group.by = 'celltype', pt.size = 0.5, label = TRUE)
plot <- plot + ggtitle(paste("spread:", spread))
# 将图片添加到列表中
plot_list[[i]] <- plot
}
# 使用 patchwork 的 wrap_plots 函数将所有图片组合成两列
final_plot <- wrap_plots(plot_list, ncol = 2) & theme(plot.title = element_text(size = 16, face = "bold"))
final_plot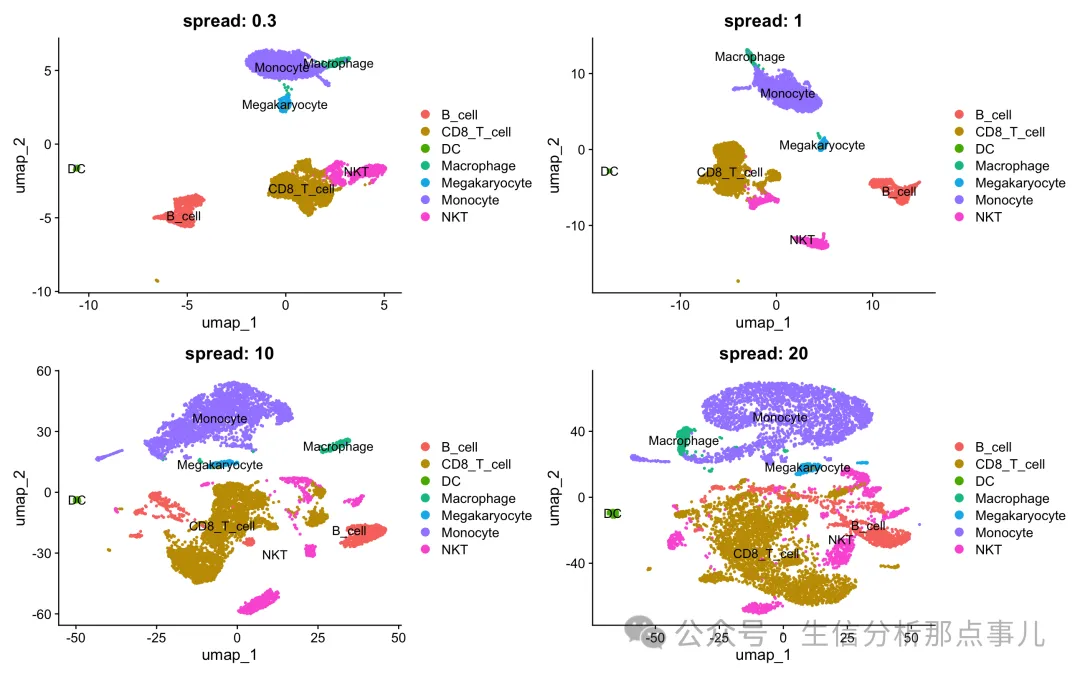 可以看到:spread越小,相似细胞cluster会更聚集:比如0.3的时候CD8和NKT挨的很近,而spread是1的时候,NKT中的一个cluster会和CD8离得很远。所以如果想让肉眼可见离群的cluster少一点,就spread小一点。
可以看到:spread越小,相似细胞cluster会更聚集:比如0.3的时候CD8和NKT挨的很近,而spread是1的时候,NKT中的一个cluster会和CD8离得很远。所以如果想让肉眼可见离群的cluster少一点,就spread小一点。
 最后附冷笑话一则:
最后附冷笑话一则:
有一天RunUMAP遇到RunTSNE,便问:兄弟你怎么变成这样了?RunTSNE回答:因为我spread开了。
总结
- PCA dims: 越小越聚集;越大越分开。
n.neighbors:越小越枝丫,越大越圆润。min.dist: 越小越碎,越大越越圆润。spread:越小越收缩,越大越胖。